
骨髄異形成症候群(Myelodysplastic Syndromes; 以下MDS)は造血幹細胞に遺伝子異常が生じることによりクローンが出現・増大する難治性の造血器腫瘍である。1近年、爆発する技術革新により網羅的な遺伝子解析が可能となり、がん化を直接促進する様々な標的が同定されている2, 3。MDSは非常に多様性に富んでいる。再生不良性貧血のような良性疾患と重複する性質を有するものから、白血病の前段階に相当するような悪性腫瘍の特徴を有するものまでを含む疾患である。同一の厳格な基準を用いた場合においても治療反応性や有害事象の重篤度、発症頻度などを事前に予測することが困難である。また、高齢者に多く発症するため今後の社会の高齢化に伴って患者数がさらに増加する。そのため、患者ごとに正確な予後予測を行う個別化医療の確立が求められている。副作用が少なく有効な治療選択が期待される。
人工知能、機械学習やビッグデータという言葉は様々なメディアで耳にすることが多いが、医療現場でどのように活用できるのかいまだ不明である。今後、これらの革新的技術を医療分野、特に血液内科の臨床レベルに落とし込み遺伝子異常の臨床応用をメインテーマに研究を遂行している。
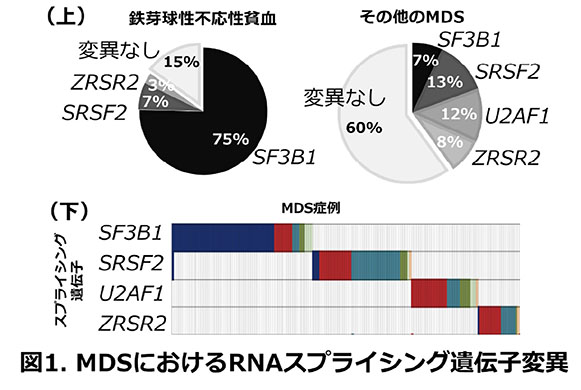
MDSには平均して約10個の体細胞変異が存在するが、腫瘍化に直接かかわるような遺伝子異常が腫瘍細胞に複数個共存しており、同一患者の腫瘍内における不均一性の存在を考慮すると、個々の遺伝子変異の腫瘍発症への役割を同定するのは容易ではない。クローン進化の多様性はMDSの特徴であるが、クローン構造が遺伝子 変異の獲得とともに経時的にどのような変化を及ぼし腫瘍化するのか詳細は不明であった。
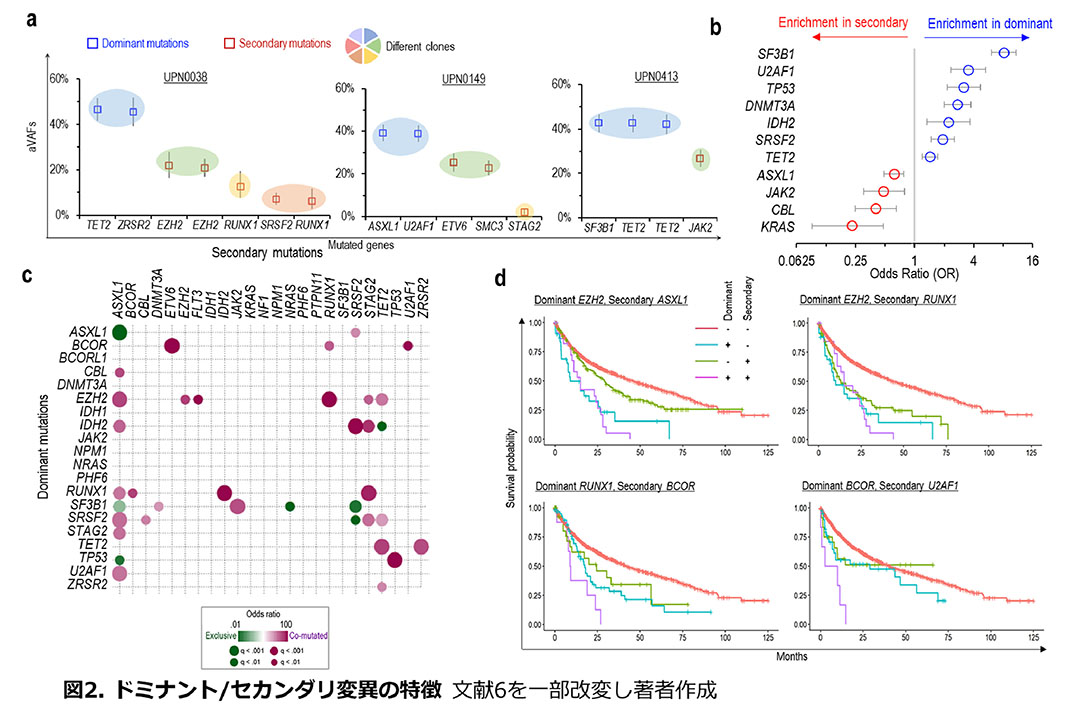
パイクローンと呼ばれる単一細胞シークエンス技術を用いて検証がされているクローン推定法を用いて1,809症例のMDSに対してクローン推定を行い、遺伝子変異の序列に関しての解析をおこなった6。次世代シークエンサーによる標的シークエンシングを行うことで3,971個の変異が同定され、これらがクローン構造の推定に用いられた。最も大きなクローンの形成に関わる変異をドミナント変異、それ以外の小クローンに関わる変異をセカンダリ変異と定義し、それぞれの変異がクローン進展とともにどのように腫瘍進展に関わるのか解析を行った。
結果として、i)サブクローンに変異を生じやすい遺伝子が存在する、ii) 初期と後期の遺伝子異常には特定のルールが存在する、iii)サブクローンにおきる遺伝子変異が予後を変える, ことを発見した(以下図2)。
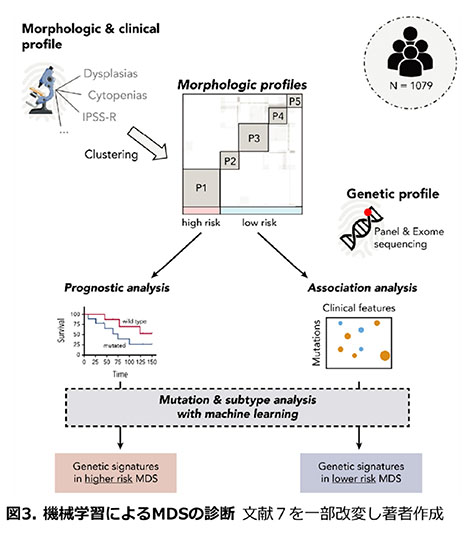
造血器疾患における遺伝子解析の依頼は随時受け付けています。倫理委員会の書類が必要であれば共有できます。検体の送付先など詳細は研究責任者の永田安伸(y-nagata@nms.ac.jp)までご連絡いただけますと幸いです。大学院生、日本国内で研究を行いたい留学生など随時募集中です。
1. Nagata Y, Maciejewski JP. The functional mechanisms of mutations in myelodysplastic syndrome. Leukemia. 2019; 33(12):2779–2794.
2. Nagata Y, Kontani K, Enami T, Kataoka K, Ishii R, Totoki Y, et al. Variegated RHOA mutations in adult T-cell leukemia/lymphoma. Blood. 2016; 127(5): 596-604.
3. Nagata Y, Narumi S, Guan Y, Przychodzen BP, Hirsch CM, Makishima H, et al. Germline loss-of-function SAMD9 and SAMD9L alterations in adult myelodysplastic syndromes. Blood. 2018;132(21): 2309-2313.
4. Yoshida K, Sanada M, Shiraishi Y, Nowak D, Nagata Y, Yamamoto R, et al. Frequent pathway mutations of splicing machinery in myelodysplasia. Nature. 2011; 478(7367): 64-69.
5. Haferlach T, Nagata Y, Grossmann V, Okuno Y, Bacher U, Nagae G, et al. Landscape of genetic lesions in 944 patients with myelodysplastic syndromes. Leukemia. 2014; 28(2): 241-247.
6. Nagata Y, Makishima H, Kerr CM, Przychodzen BP, Aly M, Goyal A, et al. Invariant patterns of clonal succession determine specific clinical features of myelodysplastic syndromes. Nat Commun. 2019; 10(1): 5386.
7. Nagata Y, Zhao R, Awada H, Kerr CM, Mirzaev I, Kongkiatkamon S, et al. Machine learning demonstrates that somatic mutations imprint invariant morphologic features in myelodysplastic syndromes. Blood. 2020; 136(20):2249-2262.